10.4 微生物数据完整注释问题
微生物数据分析中,经常会将数据折叠成门纲目科属种不同级别进行下游分析。但是受限于数据库注释的不规范,个别菌种可能存在下级注释同名,而上级注释不同名的情况。这种情况会导致EMP_collapse函数在属级别数据折叠中,错误的将不同的菌合并在一起。如下:
| Family | Genus | Species |
|---|---|---|
| Lachnospiraceae | Ruminococcus | unclassified |
| Ruminococcaceae | Ruminococcus | unclassified |
| Oscillospiraceae | Ruminococcus | unclassified |
因此,EasyMultiProfiler提供了两种微生物注释方式:完整层级注释和单一层级注释。用户可以根据需求选择合适的方式。完整层级注释在注释上更为严谨,适合进行不涉及具体细菌名称的分析和绘图(如α多样性、β多样性分析)。这种情况下,推荐直接使用完整层级注释名称。如果涉及具体细菌名称的绘图,建议先使用ggsave保存图形,再手动调整标签和注释,以提高图形的可读性。单一层级注释则在绝大多数情况下能够满足需求,并且不会影响可视化效果。
🏷️示例1:
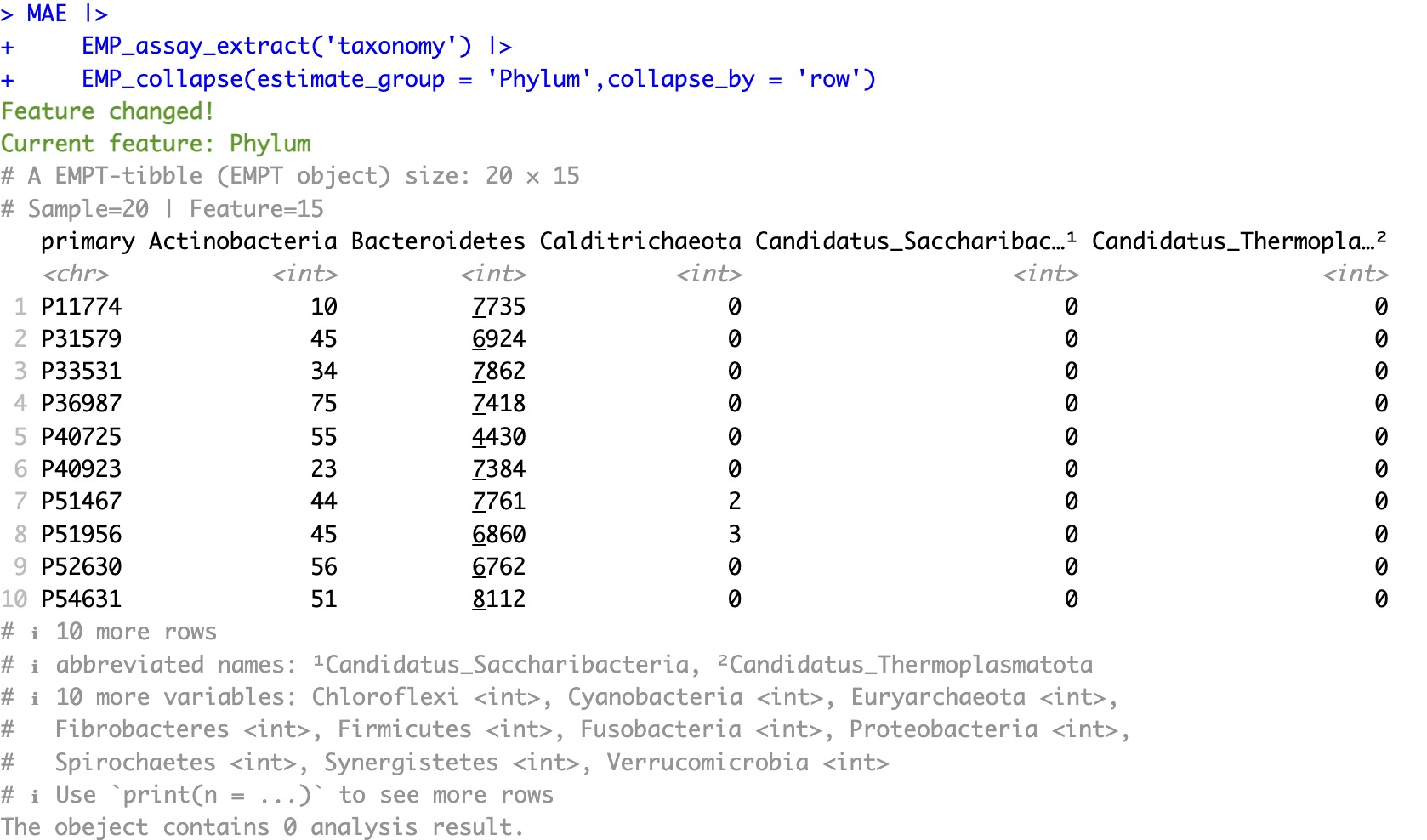
原始注释为单一层级注释:
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_collapse(estimate_group = 'Phylum',collapse_by = 'row')
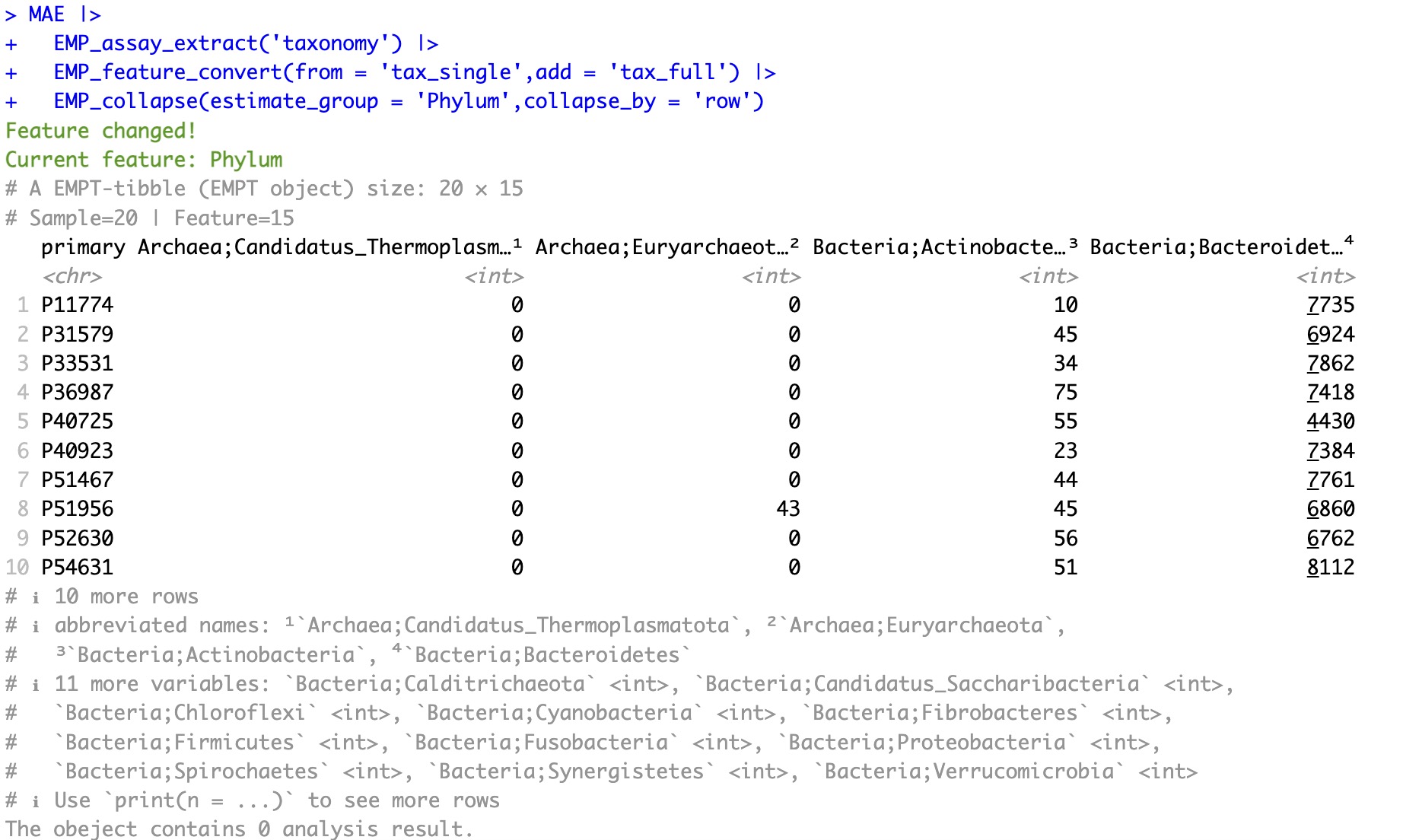
转换为完整注释:
注意:
此函数仅能在微生物数据折叠前使用。
此函数仅能在微生物数据折叠前使用。
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_feature_convert(from = 'tax_single',add = 'tax_full') |>
EMP_collapse(estimate_group = 'Phylum',collapse_by = 'row')
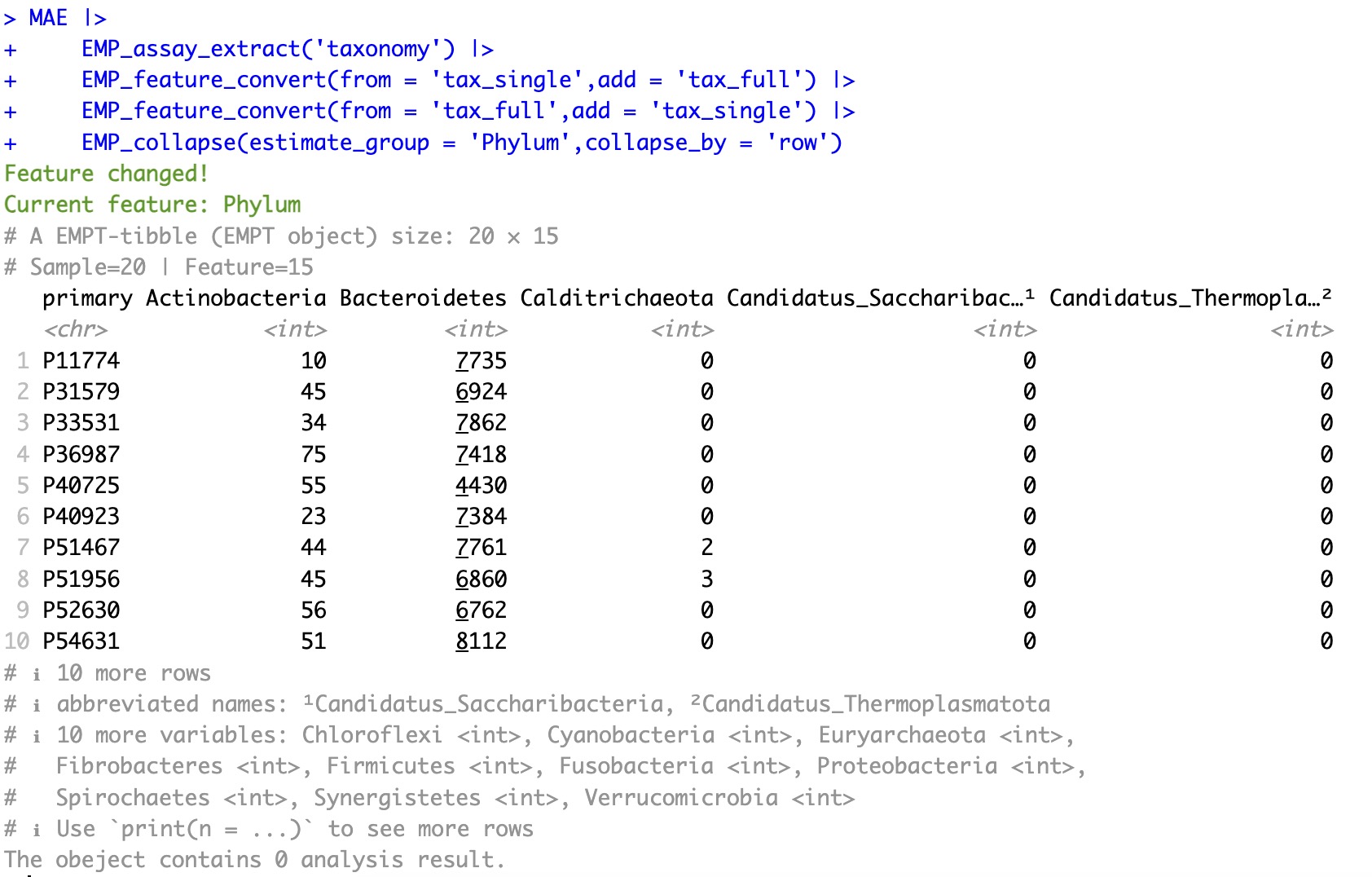
将完整注释转换回单一层级注释:
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_feature_convert(from = 'tax_single',add = 'tax_full') |>
EMP_feature_convert(from = 'tax_full',add = 'tax_single') |>
EMP_collapse(estimate_group = 'Phylum',collapse_by = 'row')
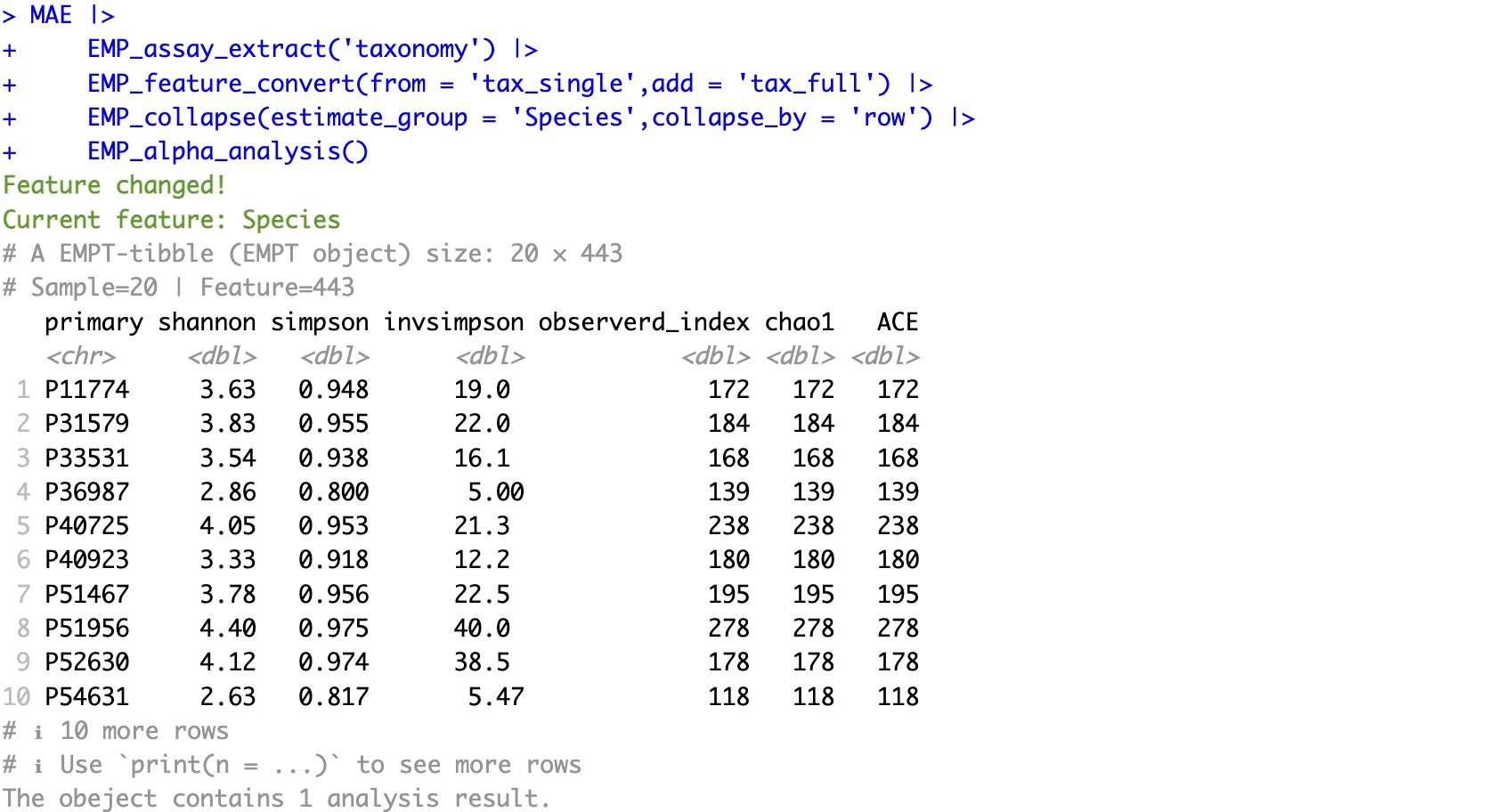
🏷️示例2: 完整注释后进行多样性分析
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_feature_convert(from = 'tax_single',add = 'tax_full') |>
EMP_collapse(estimate_group = 'Species',collapse_by = 'row') |>
EMP_alpha_analysis()